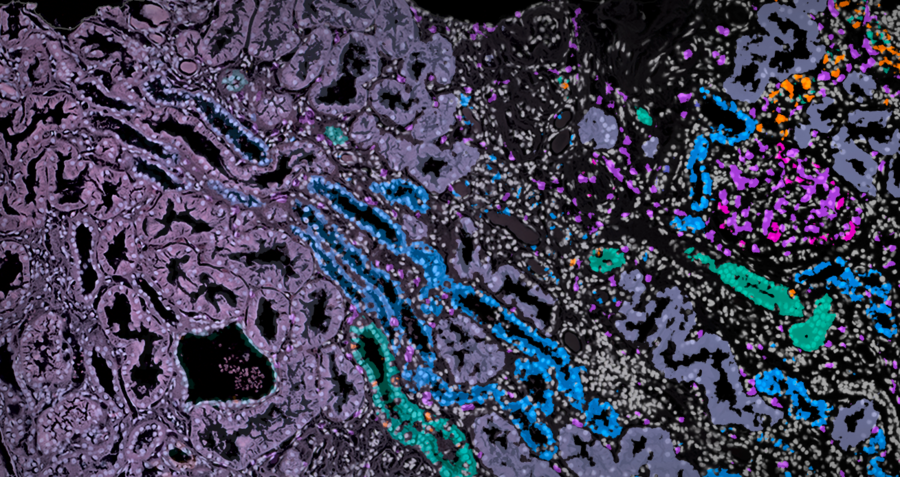
Mathematische Methoden machen komplexe Zellstrukturen in hochaufgelösten Nierendaten sichtbar © Katherine Benjamin/University of Oxford
Weltweit wird prognostiziert, dass Nierenerkrankungen bis 2040 die fünfthäufigste Todesursache sein werden, und etwa 850 Millionen Menschen leben mit einer chronischen Nierenerkrankung. Diese Krankheit beruht häufig auf einer autoimmunen Schädigung der Filtrationseinheiten der Niere, der so genannten Glomeruli, die bei Lupus auftreten kann. Von dieser Krankheit sind Frauen und nicht-weiße ethnische Gruppen, die in der Forschung häufig unterrepräsentiert sind, überproportional betroffen. Die Behandlungsmöglichkeiten sind begrenzt, können lebensbedrohliche Nebenwirkungen haben und führen oft nicht zu einer Verlangsamung der Krankheit, die dann in ein Endstadium übergehen kann, in dem eine Dialyse oder eine Nierentransplantation erforderlich ist.
Es besteht dringender Bedarf, sicherere, wirksamere und personalisierte Behandlungen für diese Nierenkrankheiten zu entwickeln. Gemeinsam mit Mathematikerinnen und Mathematikern aus Oxford entwickelte Heather Harrington, Professorin für Mathematik an der TU Dresden, der Universität Oxford und Direktorin am Max-Planck-Institut für molekulare Zellbiologie und Genetik (MPI-CBG) in Dresden und dem Zentrum für Systembiologie Dresden (CSBD) neue mathematische Ansätze zur Quantifizierung von Daten aus neuen experimentellen Technologien.
Seit etwa zehn Jahren gibt es Verfahren, die es Forschenden ermöglichen, die Genexpression in einzelnen Zellen zu verstehen, nachdem sie diese aus einer Nierenprobe herausgelöst haben. Erst seit kurzem gibt es neue Techniken, die diese Zellsignaturen in einem Gewebekontext sichtbar machen. Das heißt, die Zellen können als Teil ihrer lokalen Umgebung verstanden werden und sie zeigen, wie sich Zellen bewegen und sich gegenseitig Signale geben. Dieser Ansatz ist für Autoimmunerkrankungen der Niere wie Lupus sehr interessant, denn die Niere ist ein komplexes Gebilde mit mehr als 30 verschiedenen Zelltypen, zu denen bei Lupus eine Vielzahl von Immunzellen hinzukommen. Wie diese Interaktionen zu einer Schädigung der Niere führen, ist jedoch noch nicht gut erforscht.
Aneesha Bhandari, betreut von der Oxford Forschungsleiterin und Nephrologin Dr. Katherine Bull, setzte diese neuen räumlichen Methoden ein, um die Lupus-Nierenerkrankung zu untersuchen, wobei Katherine und Aneesha jedoch auf ein Problem stießen: Die räumlichen Informationen haben eine noch nie dagewesene subzelluläre Auflösung, sind jedoch sehr spärlich und verrauscht. Daher war es schwierig, mit diesen neuen Methoden zu entscheiden, welche Art von Zellen sie gefunden hatten und wo die Grenze jeder Zelle liegt. Die Forscherinnen wussten, dass es einen Weg geben musste, die Informationen über die lokale Expression dynamisch zu nutzen, um dies zu verbessern, aber wie? Ein Gespräch mit den Mathematikprofessorinnen Heather Harrington und Ulrike Tillmann und ihrer Studentin Katherine Benjamin ergab eine elegante Lösung. Ihre Forschung im Bereich der topologischen Datenanalyse identifiziert räumliche Muster in Daten über verschiedene Parameter hinweg. Gemeinsam entwickelten die Teams eine neue Methode, TopACT, um diese Mathematik auf die räumlichen Daten von Nieren anzuwenden. Sie waren in der Lage, verborgene Muster in der Lupus-Niere aufzudecken, wobei Immunzellen die glomerulären Regionen umkreisten. Der Ansatz funktioniert auf einer Reihe von räumlichen Plattformen, was insofern wichtig ist, als sich diese Technologien schnell weiterentwickeln und in Zukunft auch auf dreidimensionale Daten angewendet werden könnten.
Katherine Bull erklärt: „Dies ist eine wirklich interdisziplinäre Zusammenarbeit zwischen Mathematik und Biologie, die es uns ermöglicht, feinste Details und verborgene Entzündungsmuster in der Niere bei Lupus zu erkennen. Diese Werkzeuge sind ein wichtiger Schritt auf dem Weg zu einer gezielteren Behandlung dieser komplexen Krankheit.“
Heather Harington ergänzt: „Wir sind begeistert, dass wir für diese neue räumliche Transkriptomik-Technologie neuartige mathematische Ansätze entwickeln mussten, die auf modernster topologischer Datenanalyse aufbauen. In dieser Arbeit identifizierten wir die räumlichen Positionen von dünn verteilten Zellen und stellten dann die Hypothese auf, dass es sich um einen Ring von Immunzellen im Nierengewebe von Mäusen handelt, was experimentell bestätigt werden konnte. Der Versuch, diese komplexen biologischen Daten zu verstehen, bietet interessante und anspruchsvolle mathematische Möglichkeiten.“
Benjamin, K., Bhandari, A., Kepple, J.D. et al. Multiscale topology classifies cells in subcellular spatial transcriptomics. Nature (2024). https://doi.org/10.1038/s41586-024-07563-1